Características, estrutura e funções de proteínas SSB
- 4038
- 1021
- Ernesto Bruen
As Proteínas SSB o Proteínas de ligação ao DNA banda simples (do inglês "svirilha-sDNA de trand bProteínas de Inding “), Eles são proteínas responsáveis por estabilizar, proteger e manter temporariamente.
A informação genética de um organismo é protegida e codificada em DNA de banda dupla. Para que isso possa ser traduzido e replicado, é necessário que ele relaxe e desemprego e esteja nesse processo em que as proteínas SSB participam.
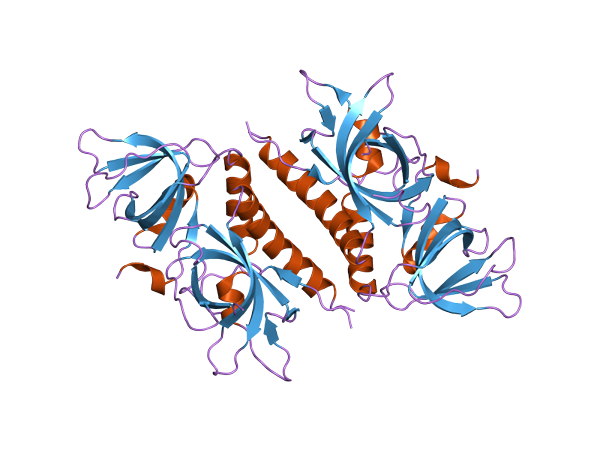
 32 Fragmento KDA (RPA32) de uma subunidade da proteína de replicação (Fonte: Jawahar Swaminathan e funcionários do MSD no Instituto Europeu de Bioinformática [Domínio Público] via Wikimedia Commons via Wikimedia)
32 Fragmento KDA (RPA32) de uma subunidade da proteína de replicação (Fonte: Jawahar Swaminathan e funcionários do MSD no Instituto Europeu de Bioinformática [Domínio Público] via Wikimedia Commons via Wikimedia) Essas proteínas são cozidas cooperativamente com outros monômeros diferentes que participam da estabilização de seu DNA e são encontrados em procariontes e eucariotos.
Proteínas SSB de Escherichia coli (ECSSB), foram as primeiras proteínas descritas desse tipo. Estes foram caracterizados por funcional e estruturalmente e, desde a descoberta, foram usados como modelo de estudo para esse tipo de proteína.
Os organismos eucarióticos têm proteínas semelhantes às proteínas SSB de bactérias, mas em eucariotos estes são conhecidos como proteínas RPA ou proteínas de replicação A (do inglês Replicação de proteínas A) que eles são funcionalmente semelhantes ao SSB.
Desde sua descoberta, modelos bioquímicos-funcionais computacionais têm sido usados para o estudo de interações entre proteínas SSB e DNA simples de cadeia, a fim de elucidar sua função nos processos essenciais do genoma de diferentes organismos.
[TOC]
Caracteristicas
Esse tipo de proteína é encontrado em todos os reinos da vida e, embora compartilhem as mesmas propriedades funcionais, elas são estruturalmente diferentes, especialmente em termos de suas mudanças conformacionais, que parecem ser específicas para cada tipo de proteína SSB.
Pode atendê -lo: Sistema ABO: incompatibilidade, herança e provaObservou -se que todas essas proteínas compartilham um domínio preservado envolvido na união com o DNA simples da banda e que é conhecido como domínio de união de oligonucleotídeos/ oligossacarídeos (é encontrado na bibliografia como domínio Ob).
Proteínas SSB de bactérias termofílicas, como Thermus aquaticus Eles têm características notáveis, uma vez que têm dois domínios OB em cada subunidade, enquanto a maioria das bactérias tem apenas uma delas em cada subunidade.
A maioria das proteínas SSB se liga não específica ao DNA da banda simples. No entanto, a união de cada SSB depende de sua estrutura, grau de cooperativa, nível de oligomerização e várias condições ambientais.
A concentração de íons divalentes de magnésio, a concentração de sais, o pH, a temperatura, a presença de poliaminas, espermidina e esperma, são algumas das condições ambientais estudadas Em vitro que mais afetam a atividade das proteínas SSB.
Estrutura
As bactérias têm proteínas SSB homo-tetraméricas, e cada subunidade tem um único domínio da União OB. Pelo contrário, as proteínas SSB virais, especialmente a de muitos bacteriófagos, são geralmente mono ou dimétricas.
No final do terminal N, as proteínas SSB têm o domínio da junção de DNA, enquanto sua extremidade C-terminal é composta por nove aminoácidos preservados encarregados das interações proteína-proteína.
Três resíduos de triptofano nas posições 40, 54 e 88 são os resíduos responsáveis pela interação com o DNA nos domínios da União. Estes mediam não apenas a estabilização da interação DNA-proteína, mas também o recrutamento das outras subunidades de proteínas.
Proteína SSB de E. coli Foi modelado em estudos computacionais e foi determinado que possui uma estrutura tetramérica de 74 kDa e que se junta ao DNA da banda simples graças à interação cooperativa de diferentes subunidades do tipo SSB.
Pode atendê -lo: vermelho de fenol: características, preparação, aplicaçõesArchaeas também têm proteínas SSB. Estes são monoméricos e têm apenas um domínio de DNA ou OB de domínio.
Nos eucariotos, as proteínas RPA são, estruturalmente, mais complexas: são compostas por um heterotrômer (de três subunidades diferentes) conhecido como RPA70, RPA32 e RPA14.
Eles têm pelo menos seis domínios de oligonucleotídeos/oligossacarídeos, embora hoje apenas quatro desses sites sejam conhecidos com precisão: três na subunidade RPA70 e uma sala que reside na subunidade RPA32.
Funções
As proteínas SSB têm funções -chave na manutenção, embalagem e organização do genoma, protegendo e estabilizando os fios de DNA de cadeia simples no momento em que são expostos pela ação de outras enzimas.
É importante ter em mente que essas proteínas não são as proteínas responsáveis por desenrolar e abrir fios de DNA. Sua função é restrita apenas a estabilizar o DNA quando está na condição do DNA simples da banda.
Essas proteínas SSB agem cooperativamente, uma vez que a união de uma delas facilita a união de outras proteínas (SSB ou não). Nos processos metabólicos de DNA, essas proteínas são consideradas um tipo de pioneira ou proteínas primárias.
A união dessas proteínas para o DNA tem como função principal, além de estabilizar as bandas de DNA de cadeia simples, a proteção dessas moléculas para a degradação por endonucleases do tipo V.
As proteínas do tipo SSB participam ativamente de praticamente todos os processos de DNA de organismos vivos. Tais proteínas avançam como o garfo de replicação avança e mantenha as duas cadeias de DNA dos pais separadas.
Pode atendê -lo: lipase pancreática: estrutura, funções, valores normaisExemplos
Nas bactérias, as proteínas SSB estimulam e estabilizam as funções de proteína. Esta proteína é responsável pelo reparo do DNA (reação SOS) e o processo de recombinação entre moléculas de banda complementares simples e complementares.
Os mutantes de E. coli Geneticamente manipulado para obter proteínas SSB defeituosas são rapidamente inibidas e não cumprem efetivamente suas funções na replicação, reparo e recombinação do DNA.
As proteínas do tipo RPA controlam a progressão do ciclo celular em células eucarióticas. Especificamente, acredita -se que a concentração celular da RPA4 possa ter uma influência indireta na passagem da replicação do DNA, isto é, em altas concentrações de RPA4, esse processo é inibido.
Foi sugerido que a expressão da RPA4 pode impedir a proliferação celular inibindo a replicação e desempenhando um papel na manutenção e marcação da viabilidade de células saudáveis em organismos animais.
Referências
- Anthony, e., & Lohman, T. M. (2019, fevereiro). Dinâmica de e. COLI SUPLO SINGRAND DNA LINDO (SSB) Complexos DNA-DNA. Em Seminários em Cell & Developmental Biology (Vol. 86, pp. 102-111). Academic Press.
- Beernink, h. T., & MORRICAL, S. C. (1999). RMPs: proteínas mediadoras de recombinação/replicação. Tendências em ciências bioquímicas, 24(10), 385-389.
- Bianco, p. R. (2017). A história do SSB. Progresso na biofísica e biologia molecular, 127, 111-118.
- Byrne, b. M., & Oakley, G. G. (2018, novembro). Proteína A Replicação, o laxante que mantém o DNA regular: a importância da fosforilação da RPA na manutenção da estabilidade do genoma. Em Seminários em Cell & Developmental Biology. Academic Press
- Krebs, J. E., Goldstein, e. S., & Kilpatrick, S. T. (2017). Genes de Lewin xii. Jones e Bartlett Learning.
- LECONTE, f., Serena, c., Velten, m., Custos, a., McGovern, s., Meile, j. C.,… & Pollard, P. (2007). Antecipando a replicação cromossômica PRIMAÇÃO DA FILHO: SSB Repair helicases de DNA para garfos ativos. O diário EMBO, 26(19), 4239-4251.
- « Características de amplitude das ondas, fórmulas e exercícios
- Características, estilos e estratégias de aprendizado ativo »

