Métodos de estudo de haplootype, diagnósticos, doenças
- 1785
- 104
- Melvin Mueller
A Haplootype É uma região do genoma que tende a herdar juntos através de várias gerações; Normalmente, tudo está localizado no mesmo cromossomo. Haplootipos são o produto da ligação genética e permanecem intactos durante a recombinação genética.
A palavra "haplotipo" deriva de uma combinação da palavra "haplóide" e a palavra "genótipo". "Haplóide" refere -se a células com um único conjunto de cromossomos e "genótipo" fala sobre a composição genética de um organismo.
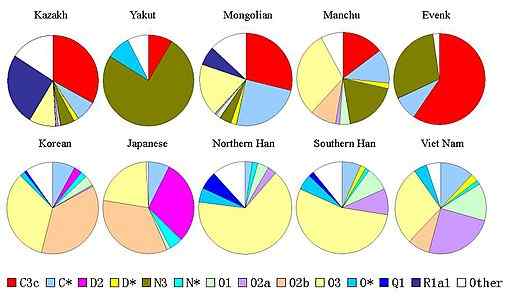
 Esquema da distribuição de haplótipos cromossômicos e em populações asiáticas (Fonte: Moogalord [domínio público] via Wikimedia Commons) Seguindo a definição, um haplótipo pode descrever alguns genes ou mais que são herdados juntos em um cromossomo de um parental, ou pode descrever um cromossomo completamente herdado de um parental, como é o caso do cromossomo e dos homens.
Esquema da distribuição de haplótipos cromossômicos e em populações asiáticas (Fonte: Moogalord [domínio público] via Wikimedia Commons) Seguindo a definição, um haplótipo pode descrever alguns genes ou mais que são herdados juntos em um cromossomo de um parental, ou pode descrever um cromossomo completamente herdado de um parental, como é o caso do cromossomo e dos homens.
Por exemplo, quando os haplótipos compartilham genes para dois caracteres fenotípicos diferentes, como cor de cabelo e cor dos olhos, indivíduos que têm gene da cor do cabelo também possuirão o outro gene para a cor dos olhos.
Haplootipos são uma das ferramentas mais usadas atualmente para o estudo da genealogia, para rastrear a origem das doenças, para caracterizar a variabilidade genética e a filogeografia de populações de diferentes tipos de seres vivos.
Existem várias ferramentas para o estudo dos haloootipos, um dos mais utilizados hoje é "Mapa do haplótipo“(Hapmap), que é uma página da web que permite determinar quais são os segmentos do genoma que são haplótipos.
[TOC]
Métodos de estudo
Haplootipos representam uma oportunidade de entender a herança dos genes e seu polimorfismo. Com a descoberta da técnica "reação em cadeia da polimerase" (PCR, inglês "Reação em cadeia da polimerase”) Ele avançou amplamente no estudo de haplótipos.
Atualmente, existem numerosas metodologias para o estudo de haplótipos, alguns dos mais destaques são:
Sequenciamento de DNA e detecção de polimorfismo de nucleotídeo único (SNPs)
O desenvolvimento de tecnologias de sequenciamento de novas gerações representou um grande salto para o estudo de haplótipos. Novas tecnologias permitem variações de até uma única base de nucleotídeos em regiões específicas de um haplótipo.
Pode atendê -lo: DNA recombinante: técnica, aplicações e fundaçõesNa bioinformática, o termo haplótipo também é usado para se referir à herança de um único polimorfismos de nucleotídeo (SNP) em sequências de DNA.
A combinação de programas de bioinformática com a detecção de haplótipos usando o sequenciamento de nova geração pode ser exatamente identificada a posição, substituição e efeito da mudança de cada base no genoma de uma população.
Microssatellites (SSRS)
Os microssatélites ou SSRs derivam seu nome dos ingleses “simplementar repetição e Repita em tandem curto". Essas são sequências de nucleotídeos curtas que são repetidas sucessivamente dentro de uma região do genoma.
É comum encontrar microssatélites dentro dos haplótipos não codificadores; portanto, através da detecção de variações no número de repetições de microssatélites, diferentes alelos podem ser observados nos haplótipos dos indivíduos.
Marcadores moleculares microssatélites foram desenvolvidos para a detecção de haplotipos intermináveis, a partir do sexato de plantas como mamão (Carica Papaya) até a detecção de doenças humanas, como anemia falciforme.
Polimorfismos dos fragmentos amplificados (AFLP)
Esta técnica combina amplificação com reações de PCR com digestão de DNA com duas enzimas de restrição diferentes. A técnica detecta locos polimórficos em haplatipos de acordo com os diferentes locais de corte na sequência de DNA.
Para ilustrar melhor a técnica, vamos imaginar três fragmentos de tecido do mesmo comprimento, mas cortados em lugares diferentes (esses fragmentos representam três fragmentos de haplótipos amplificados usando a técnica de PCR).
Na época em que o tecido é cortado, muitos pedaços de tamanho diferente serão obtidos, pois cada pano é cortado em lugares diferentes. Ao pedir os fragmentos de acordo com o tipo de tecido do qual eles vêm, podemos observar onde as diferenças entre tecidos ou haplótipos são encontrados.
Diagnóstico e doenças
Uma vantagem importante do estudo genético dos haplótipos é que estes permanecem quase intactos ou inalterados por milhares de gerações, e isso permite a identificação de ancestrais remotos e cada uma das mutações com as quais os indivíduos contribuem para o desenvolvimento de doenças.
Pode atendê -lo: variações fenotípicasHaplootipos na humanidade variam dependendo das raças e, a partir desta colher, os genes foram detectados em haplótipos que causam doenças graves em cada uma das raças humanas.
No projeto Hapmap Quatro grupos raciais estão incluídos: iorubá europeu, nigeriano, han chinês e japonês.
Dessa forma, o projeto Hapmap Ele pode cobrir os diferentes grupos de populações e rastrear a origem e a evolução de muitas das doenças hereditárias que afetam cada uma das quatro raças.
Uma das doenças que são diagnosticadas com mais frequência usando análise de haplótipos é a anemia falciforme em humanos. Esta doença é diagnosticada rastreando a frequência de haplótipos africanos em uma população.
Sendo uma doença originalmente da África, a identificação de haplótipos africanos em populações permite rastrear facilmente pessoas que possuem a mutação na sequência genética para as beta globinas de eritrócitos na forma de uma HOZ (característica da patologia).
Exemplos
Com haplótipos árvores filogenéticas que representam relações evolutivas entre cada um dos haplótipos encontrados em uma amostra de moléculas de DNA homólogas ou na mesma espécie, em uma região que tem pouca ou nenhuma recombinação é construída.
Um dos ramos mais estudados através de haplótipos é a evolução do sistema imunológico dos seres humanos. Haplootipos que codificam o receptor do tipo pedágio (um componente -chave do sistema imunológico inato) foram identificados para o genoma Neandertal e Denisovanos.
Isso lhes permite rastrear como as seqüências genéticas mudaram nas populações de humanos "modernos" em relação às sequências de haplótipo que correspondem a humanos "ancestrais".
Construindo uma rede de relações genéticas a partir do haplótipo mitocondrial.
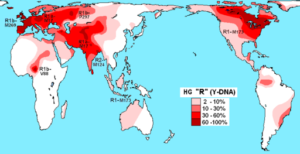 Distribuição do haplogrupo R (DNA-Y) em populações nativas (Fonte: Maucioni [CC por 3.0 (https: // CreativeCommons.Org/licenças/por/3.0)], via Wikimedia Commons) A diversidade de haplótipos é usada para rastrear e estudar a diversidade genética dos animais criados em cativeiro. Essas técnicas são usadas especialmente para espécies difíceis para monitorar a vida selvagem.
Distribuição do haplogrupo R (DNA-Y) em populações nativas (Fonte: Maucioni [CC por 3.0 (https: // CreativeCommons.Org/licenças/por/3.0)], via Wikimedia Commons) A diversidade de haplótipos é usada para rastrear e estudar a diversidade genética dos animais criados em cativeiro. Essas técnicas são usadas especialmente para espécies difíceis para monitorar a vida selvagem.
Espécies animais, como tubarões, pássaros e grandes mamíferos, como onças, elefantes, entre outros, são constantemente avaliados geneticamente através de haplótipos mitocondriais para monitorar o estado genético das populações em cativeiro.
Referências
- Bahlo, m., Stankovich, J., Velocidade, t. P., Rubio, J. P., Burfoot, r. K., & Foot, S. J. (2006). Defetendo o compartilhamento de haplótipos de genoma usando dados de haplótipo SNP ou microssatélite. Human Genetics, 119 (1-2), 38-50.
- Dannemann, m., Andrés, a. M., & Kelso, J. (2016). A introgrento dos haplótipos de neanderthal-ando denisovan contribuem para a variação adaptativa em receptores de pedágio humano. The American Journal of Human Genetics, 98 (1), 22-33.
- De Vries, H. G., Van der Meulen, M. PARA., Rozen, r., Halley, d. J., Scheffer, h., Leo, p.,… E você meerman, g. J. (mil novecentos e noventa e seis). Identidade do haplótipo entre indivíduos que compartilham um alelo de mutação CFTR. Human Genetics, 98 (3), 304-309
- Engolir, m. PARA., Leaver, a. eu., Christiansen, f. T., Witt, c. S., Abraham, l. J., & Dawkins, r. eu. (1992). Haplotipos ancestrais: Haplótipos de MHC da população de conserva. Human Immunology, 34 (4), 242-252.
- Bolsistas, m. R., Hartman, t., Hermelin, d., Landau, g. M., Rosamond, f., & Rozenberg, L. (2009, junho). Inferência de haplótipo restrito por dados de haplótipo plausível. No simpósio anual sobre correspondência de padrões combinatórios (pp. 339-352). Springer, Berlim, Heidelberg.
- Gabriel, s. B., Schaffner, s. F., Nguyen, h., Moore, J. M., Roy, J., Blumenstiel, b.,... & Liu-Cordermo, S. N. (2002). A estrutura dos blocos de haplótipo no genoma humano. Science, 296 (5576), 2225-2229.
- Consórcio Internacional HapMap. (2005). Um mapa de haplótipos do genoma humano. Nature, 437 (7063), 1299.
- Wynne, r., E selvagem, C. (2018). MithoCondrial DNA Haplótipo Diversidade e origem de tubarões de areia em cativeiro (Carcharias Taurus). Journal of Zoo and Aquarium Research, 6 (3), 74-78.
- Yoo, e. J., Tang, j., Kaslow, r. PARA., & Zhang, k. (2007). Inferência de haplótipo para dados de genótipos atuais de Abent. Bioinformatics, 23 (18), 2399-2406.
- Jovem, n. S. (2018). Anemia aplástica. The New England Journal of Medicine, 379 (17), 1643-1656.
- « Características da herança holandesa, funções de genes, degeneração
- Lentén maiores características, habitat, propriedades, cuidados »

