Funções, tipos e exemplos de enzimas de restrição
- 2459
- 120
- Shawn Leffler
As Enzimas de restrição São endonucleases usadas por certos arcos e bactérias para inibir ou "restringir" a propagação de vírus dentro. Eles são especialmente comuns em bactérias e fazem parte de seu sistema de defesa de DNA estrangeiro conhecido como sistema de restrição/modificação.
Essas enzimas catalisam o corte de DNA de banda dupla em lugares específicos, reproduzível e sem o uso de energia adicional. A maioria requer a presença de cofatores como magnésio ou outros cátions divalentes, embora alguns também exijam ATP ou S-adenosil metionina.
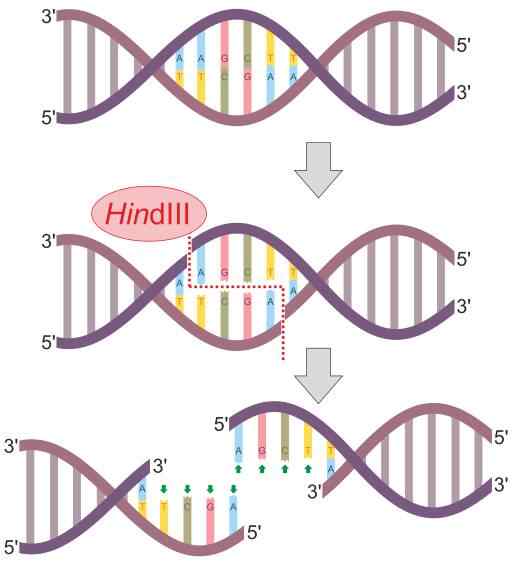
 Esquema de reação de enzima de restrição Hindii (fonte: Helixitta [CC BY-SA 4.0 (https: // CreativeCommons.Org/licenças/BY-SA/4.0)] via Wikimedia Commons)
Esquema de reação de enzima de restrição Hindii (fonte: Helixitta [CC BY-SA 4.0 (https: // CreativeCommons.Org/licenças/BY-SA/4.0)] via Wikimedia Commons) As endonucleases de restrição foram descobertas em 1978 por Daniel Nathans, Arber Werner e Hamilton Smith, que receberam o Prêmio Nobel de Medicina por sua descoberta. Seu nome geralmente deriva do organismo onde eles são observados pela primeira vez.
Tais enzimas são amplamente utilizadas no desenvolvimento de métodos de clonagem de DNA e outras estratégias de biologia molecular e engenharia genética. Suas características de reconhecimento de sequências específicas e capacidade de corte das sequências próximas aos sites de reconhecimento os tornam ferramentas poderosas em experimentação genética.
Os fragmentos gerados pelas enzimas de restrição que agiram em uma molécula de DNA específica podem ser usados para recriar um "mapa" da molécula original usando informações nos locais onde a enzima cortou o DNA.
Algumas enzimas de restrição podem ter o mesmo local de reconhecimento no DNA, mas não necessariamente o cortam da mesma maneira. Assim, existem enzimas que fazem cortes deixando romos e enzimas que cortam os extremos coesos, que têm aplicações diferentes na biologia molecular.
Atualmente, existem centenas de diferentes enzimas de restrição disponíveis comercialmente, oferecidas por diferentes casas comerciais; Essas enzimas funcionam como uma tesoura molecular "personalizada" para diferentes fins.
[TOC]
Funções
As enzimas de restrição cumprem a função oposta das polimerases, pois são hidrolisadas ou quebram a ligação éster dentro da ligação fosfodiéster entre nucleotídeos adjacentes em uma cadeia nucleotídica.
Na biologia molecular e engenharia genética, eles são ferramentas amplamente usadas para a construção de vetores de expressão e clonagem, bem como para a identificação de sequências específicas. Eles também são úteis para a construção de genomas recombinantes e têm um grande potencial biotecnológico.
Os recentes avanços na terapia genética tornam o uso atual das enzimas de restrição para a introdução de genes determinados em vetores que são veículos para o transporte de tais genes para células vivas e que provavelmente têm a capacidade de se inserir no genoma celular para realizar mudanças permanentes.
Mecanismo de ação
As enzimas de restrição podem catalisar o corte de DNA de banda dupla, embora algumas sejam capazes de reconhecer sequências simples de DNA de banda e até RNA. O corte ocorre após o reconhecimento das seqüências.
Pode atendê -lo: ciclo de glioxilato: características, reações, regulamentação, funçõesO mecanismo de ação consiste na hidrólise do vínculo de fosfodiéster entre um grupo fosfato e uma desoxirribose no esqueleto de cada DNA do DNA. Muitas das enzimas são capazes de cortar no mesmo lugar que reconhecem, enquanto outros correm entre 5 e 9 pares de bases antes ou depois do mesmo.
Normalmente, essas enzimas cortadas no final de 5 'do grupo fosfato, dando origem a fragmentos de DNA com uma extremidade fosforila de 5' e um terminal hidroxil extremidade 3 '.
Como as proteínas não entram em contato direto com o local de reconhecimento de DNA, elas devem ser translocadas com sucessivos tempos até que o local específico seja alcançado, talvez através de mecanismos "deslizantes" na vertente do DNA.
Durante o corte enzimático, o link de fosfodiéster de cada um dos fios de DNA está posicionado em um dos locais ativos das enzimas de restrição. Quando a enzima sai do local de reconhecimento e corte, o faz através de associações transitórias não específicas.
Pessoal
Atualmente, cinco tipos de enzimas de restrição são conhecidos. Em seguida, uma breve descrição de cada uma:
Enzimas de restrição do tipo I
Essas enzimas são grandes proteínas pentaméricas com três subunidades, uma de restrição, uma metilação e outra para o reconhecimento de sequências de DNA. Essas endonucleases são proteínas multifuncionais capazes de catalisar as reações de restrição e modificação, têm ATPASA e também a atividade de DNA topoisomera.
As enzimas desse tipo foram as primeiras endonucleases a serem descobertas, elas foram purificadas pela primeira vez na década de 1960 e, desde então, foram estudadas com grande profundidade.
As enzimas do tipo I não são amplamente utilizadas como uma ferramenta biotecnológica, pois o local de corte pode estar a uma distância variável de até 1.000 pares de bases sobre o site de reconhecimento, o que os torna não confiáveis em termos de reprodutibilidade experimental.
Enzimas de restrição tipo II
São enzimas compostas por homodímeros ou tetrâmeros que cortam o DNA em locais definidos entre 4 e 8 pb de comprimento. Esses locais de corte são tipicamente palindrômicos, ou seja, reconhecem sequências que são lidas da mesma maneira em ambas as direções.
Muitas das enzimas de restrição do tipo II em bactérias cortam o DNA quando reconhecem seu caráter estrangeiro, pois não possui as modificações típicas de que o próprio DNA deve ter.
Essas são enzimas de restrição mais simples, uma vez que não exigem nenhum cofator além do magnésio (mg+) para reconhecer e cortar as sequências de DNA.
A precisão das enzimas de restrição do tipo II no reconhecimento e corte de sequências simples no DNA em posições precisas as torna uma das mais usadas e indispensáveis na maioria dos ramos da biologia molecular.
Pode atendê -lo: Mutualismo: características, tipos, exemplosDentro do grupo de enzimas de restrição do tipo II, são várias subclasses classificadas de acordo com certas propriedades que são únicas para cada. A classificação dessas enzimas é realizada adicionando letras do alfabeto, de A a Z seguindo o nome da enzima.
Algumas das subclasses mais conhecidas por sua utilidade são:
Subclasse IIA
São diferentes subunidades dímeros. Eles reconhecem sequências assimétricas e são usadas como precursores ideais para a geração de enzimas de corte.
Subclasse IIB
Eles são compostos por mais um dímeros e cortam o DNA nos dois lados da sequência de reconhecimento. Eles cortam os dois fios de DNA em um intervalo de pares de bases posteriormente no local de reconhecimento.
Subclasse IIC
As enzimas desse tipo são polipeptídeos com funções de divisão e modificação dos fios de DNA. Essas enzimas cortam ambos os fios assimetricamente.
Subclasse IIE
As enzimas desta subclasse são as mais usadas em engenharia genética. Eles têm um local catalítico e geralmente exigem um efetor alestro. Essas enzimas precisam interagir com duas cópias de sua sequência de reconhecimento para fazer um corte eficiente. Dentro desta subclasse estão Ecorii e Ecori enzimas.
Enzimas de restrição tipo III
A endonucleases de restrição do tipo III são compostas apenas por duas subunidades, uma é responsável pelo reconhecimento e modificação do DNA, enquanto o outro é responsável pelo corte da sequência.
Essas enzimas requerem dois cofatores para sua operação: ATP e magnésio. As enzimas de restrição desse tipo têm dois sites de reconhecimento assimétricos, translocate o DNA de maneira dependente de ATP e corte -o entre 20 a 30 pb adjacentes ao local de reconhecimento.
Enzimas de restrição do tipo IV
As enzimas tipo IV são fáceis de identificar, pois cortam o DNA com marcas de metilação, são compostas por várias subunidades diferentes responsáveis por reconhecer e cortar a sequência de DNA. Essas enzimas usam cofatores GTP e magnésio divalente.
Locais de corte específicos incluem cadeias de nucleotídeos com resíduos de citosina metilados ou hidroximitados em um ou ambos os fios de ácidos nucleicos.
Enzimas de restrição do tipo V
Esta classificação Grupos. As enzimas Cas usam um guia sintetizado de fita de RNA para que reconheçam e ataquem organismos invasores.
As enzimas classificadas como tipo V são polipeptídeos estruturados pelas enzimas tipo I, II e II. Eles podem cortar seções de DNA de quase qualquer organismo e com uma grande variedade de comprimento. Sua flexibilidade e facilidade de emprego tornam essas enzimas uma das ferramentas mais usadas em engenharia genética atualmente juntamente com enzimas tipo II.
Pode atendê -lo: PropionibacteriumExemplos
As enzimas de restrição têm sido usadas para a detecção de polimorfismos de DNA, especialmente em estudos de genética populacional e estudos evolutivos usando DNA mitocondrial, a fim de obter informações sobre taxas de substituição de nucleotídeos.
Atualmente, os vetores usados para a transformação de bactérias com vários propósitos têm locais de multiclonagem, onde os locais de reconhecimento para enzimas de restrição múltiplas são encontrados.
Entre essas enzimas, as mais populares estão Eco, II, III, IV e V, obtidas e descritas pela primeira vez de E. coli; Hindiii, de H. influenzae e Bamhi de B. Amiloliquefaciens.
Referências
- Bikle, t. PARA., & Kruger, D. H. (1993). Biologia da restrição de DNA. Revisões microbiológicas, 57(2), 434-450.
- Boyaval, p., Moineau, s., Romero, d. PARA., & Horvath, P. (2007). O CRISPR fornece aquisição contra vírus em procariontes. Ciência, 315(Março), 1709-1713.
- Goodsell, d. (2002). A perspectiva molecular: endonucleases de restrição. Células -tronco Fundamentos de medicina do câncer, vinte, 190-191.
- Halford, s. E. (2001). Pular, pular e loop por enzimas de restrição. Transações da Sociedade Bioquímica, 29, 363-373.
- Jeltsch, a. (2003). Manutenção da identidade de espécies e controle da especiação de bactérias: uma nova função para sistemas de restrição/modificação? Gene, 317, 13-16.
- Krebs, J., Goldstein, e., & Kilpatrick, S. (2018). Genes de Lewin xii (12 ed.). Burlington, Massachusetts: Jones e Bartlett Learning.
- Li, e., Pão, s., Zhang, e., Ren, m., Feng, m., Peng, n.,... ela, q. (2015). Aproveitando os sistemas CRISPR-CAS Tipo I e Tipo III para edição de genoma. Pesquisa de ácidos nucleicos, 1-12.
- Loenen, w. PARA. M., Dryden, d. T. F., Raleight, e. PARA., & Wilson, G. G. (2013). Enzimas de restrição do tipo I e seus parentes. Pesquisa de ácidos nucleicos, 1-25.
- Nathans, d., & Smith, H. QUALQUER. (1975). Endonucleases de restrição na análise e reestruturação de moléculas de DNA. Annu. Rev. Biochem., 273-293.
- Nei, m., & Tajima, f. (1981). Polimorfismo de DNA detectável por endonucleases de restrição. Genética, 145-163.
- Pingoud, a., Fuxreiter, m., Pingoud, v., & Wende, w. (2005). Ciências da Vida Celular e Molecular Tipo II Endonucleases: Estrutura e Mecanismo. Ciências da vida celular e molecular da CMLS, 62, 685-707.
- Roberts, r. (2005). Como as enzimas de restrição se tornaram os cavalos de trabalho da biologia molecular. Pnas, 102(17), 5905-5908.
- Roberts, r. J., & Murray, K. (1976). Endonucleases de restrição. Revisões críticas em bioquímica, (Novembro), 123-164.
- Stoddard, b. eu. (2005). Homing endonuclease Estrutura e função. Revisões trimestrais de biofísica, 1-47.
- TOCK, m. R., & Dryden, D. T. F. (2005). A biologia da restrição e anti-restauração. Opinião atual em microbiologia, 8, 466-472. https: // doi.org/10.1016/j.meu B.2005.06.003
- Wilson, g. G., & Murray, n. E. (1991). Sistemas de restrição e modificação. Annu. Rev. Genet., 25, 585-627.
- Wu, z., & MOU, K. (2016). Insights genômicos sobre a virulência de Campylobacter jejuni e genética populacional. Infec. Dis. Trad. Med., 2(3), 109-19.
- Yuan, r. (1981). Estrutura e mecanismo de endonucleases de restrição multifuncional. Annu. Rev. Biochem., cinquenta, 285-315.
- « Geração de características, autores e obras de 1914
- Propriedades e exemplos de eventos mutuamente exclusivos »

