Mecanismos de replicação de DNA, em procariontes e eucariotos
- 3952
- 647
- Terrence King IV
O Replicação de DNA (Ácido desoxirribonucleico) Consiste em copiar o genoma, ou seja, todas as informações genéticas contidas no DNA de um organismo, para produzir duas cópias idênticas. O genoma tem as informações necessárias para construir um organismo completo.
Antes da divisão celular, a replicação do DNA ocorre. Através da meiose, os gametas para reprodução sexual ocorrem. Através da mitose, ocorre a substituição celular (por exemplo, pele e sangue) e desenvolvimento (por exemplo, tecidos e órgãos).
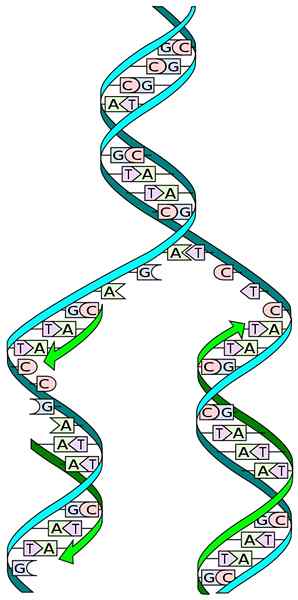
 Fonte: I, MadPrime [CC0]
Fonte: I, MadPrime [CC0] O conhecimento da estrutura de DNA nos permite entender a maneira como sua replicação ocorre. A estrutura do DNA consiste em uma hélice dupla, composta por duas cadeias antipaalais de nucleotídeos sucessivos, cujas bases de nitrogênio são complementadas especificamente.
Durante a replicação, cada uma das correntes da cadeia de DNA dupla atua como um molde para a biossíntese de uma nova cadeia. As duas cadeias recém -sintetizadas têm bases complementares às bases da cadeia de mofo: adenina (a) com timina (t) e citosina (c) com guanina (g).
Várias enzimas e proteínas participam da replicação do DNA. Por exemplo, abrir a hélice de DNA duplo, manter o DNA aberto e adicionar desoxirribonucleosidos-5'-trifosfato (DNTP) para formar a nova cadeia.
[TOC]
A replicação do DNA é semi -conservadora
Com base na estrutura do DNA, Watson e Crick propuseram que a replicação do DNA ocorra de maneira semi -contratante. Isso foi demonstrado por Meselson e Stahl marcando o DNA de Escherichia coli Com o pesado isótopo do nitrogênio, quinzeN, seguindo para várias gerações o padrão de distribuição em um meio de cultura com nitrogênio leve, 14N.
Messelson e Stahl descobriram que, na primeira geração, as duas moléculas de DNA das duas filhas tinham, cada molécula marcada com uma corrente com o isótopo de nitrogênio pesado e outro com o isótopo leve. Ao contrário da molécula de DNA dos pais, que tinha as duas correntes marcadas com o isótopo pesado, quinzeN.
Na segunda geração, 50% das moléculas de DNA eram como as da primeira geração, e os outros 50% tinham apenas nitrogênio leve. A interpretação desse resultado é que a hélice da dupla filha tem uma cadeia de pais (que funciona como um molde) e uma nova cadeia.
O mecanismo de replicação semi -contratante implica na separação de cadeias de DNA e acasalamento de bases complementares por meio de nucleotídeos sucessivos, produzindo duas filhas de filhas duplas filhas.
Replicação em baterias
Início da replicação do DNA em bactérias
O DNA de bactérias consiste em um cromossomo circular e possui apenas um local de origem da replicação. A partir deste site, a biossíntese das duas filhas acontece bidirecionalmente, formando dois garfos de replicação que se movem em direções opostas à origem. No final, os garfos estão, completando a replicação.
A replicação começa com a união das proteínas DNAA no local de origem. Essas proteínas, por sua vez, formam um complexo. Então as proteínas HU e IHF estão ligadas.
Em seguida, as proteínas DNAC são unidas, que fazem as helicases do DNA se juntarem. Isso ajuda a relaxar o DNA e quebrar as ligações de hidrogênio, formadas em pares de bases. Então, as duas cadeias se separam ainda mais, formando duas correntes simples.
Pode atendê -lo: o que é permutação cromossômica?Topoisomerase II, ou DNA Girasa, se move na frente do DNA Heliciety. As proteínas de ligação ao DNA de cadeia única (SSB) mantêm as cadeias de DNA separadas. Assim, a biossíntese da cadeia filha pode começar.
Biossíntese de cadeias de DNA de filhas em bactérias
A enzima prima é responsável por sintetizar cadeias de RNA curtas chamadas primers, que têm 10 a 15 nucleotídeos de comprimento. A polimerase de DNA começa a adicionar 5'-trifosfato (DNTPS) ao 3'-OH do açúcar de priming, após o que a corrente continua a crescer pelo mesmo fim.
Como as cadeias de DNA são antiparalelas, um primer é sintetizado na corrente de guia e muitos iniciadores na cadeia tardia. Por causa disso, a biossíntese da cadeia tardia é descontínua. Embora as cadeias de DNA sejam antiparalelas, o garfo de replicação se move em uma direção.
A polimerase de DNA é responsável pela formação de ligações covalentes entre os nucleotídeos adjacentes das cadeias recém -sintetizadas, na direção 5'®3 '. Em E. coli, Existem cinco DNA da polimerase: polimerases de DNA I e III realizam replicação de DNA; e as polimerases de DNA II, IV e V são responsáveis por replicar e replicar o DNA danificado.
A maior parte da replicação é feita pela DNA polimerase III, que é uma holoenzima que possui 10 subunidades diferentes com várias funções na replicação do DNA. Por exemplo, a subunidade alfa é responsável por fazer títulos entre nucleotídeos.
Um complexo enzimático é responsável pela replicação do DNA em bactérias
A helicase de DNA e prima é unida para formar um complexo chamado Primosoma. Isso se move ao longo do DNA, agindo de maneira coordenada para separar as duas cadeias parentais, sintetizando os iniciadores a cada intervalo de certo intervalo na cadeia tardia.
O primaossomo se liga fisicamente à DNA polimerase III e forma o replisome. Duas polimerases de DNA III são responsáveis por replicar o DNA do guia e as correntes atrasadas. Com relação à DNA polimerase III, a cadeia tardia forma um loop, o que permite que a adição de nucleotídeos a essa cadeia aconteça na mesma direção da cadeia guia.
A adição de nucleotídeos à cadeia guia é contínua. Enquanto está no atraso, é descontínuo. São formados fragmentos de 150 nucleotídeos de comprimento, chamados fragmentos de Okazaki.
A atividade da exonuclease 5 ' -> 3' da polimerase I da DNA é responsável por eliminar os primers e o preenchimento, adicionando nucleotídeos. Uma enzima ligase sela as lacunas entre fragmentos. A replicação termina quando os dois Hoquillas de replicação estão em uma sequência de conclusão.
A proteína que você se liga à sequência de terminação, interrompendo o movimento do garfo de replicação. Topoisomerase II permite a separação dos dois cromossomos.
Os imoxiribonucleotídeos trifosfatos são usados pela DNA polimerase
O trifosfato de dexinucleosídeo (DNTP) contém três grupos de fosfato unidos para carbono 5 'de desoxirribosa. Os DNTPs (DATP, DTTP, DGTP e DCTP) juntam -se à cadeia de molde após a regra AT/GC.
Pode atendê -lo: PlesiomorphyA polimerase de DNA catalisa a seguinte reação: o grupo hidroxila (-OH) 3 'do nucleotídeo da cadeia de cultivo reage com o fosfato alfa do DNTP de entrada, liberando pirofosfato inorgânico (PPI) (PPI). A hidrólise do PPI produz energia para a formação da ligação covalente, ou ligação fosfodiéster, entre nucleotídeos da cadeia de crescimento.
Mecanismos que garantem a fidelidade de replicação do DNA
Durante a replicação do DNA, a DNA polimerase III comete um erro por 100 milhões de nucleotídeos. Embora a probabilidade de erro seja muito baixa, existem mecanismos que garantem a fidelidade na replicação do DNA. Esses mecanismos são:
1) estabilidade na base apar. A energia de ligação de hidrogênio entre AT/GC é maior do que em pares de bases errôneos.
2) Estrutura do local ativo da polimerase de DNA. A polimerase de DNA catalisa preferencialmente nucleotídeos com bases corretas na cadeia oposta. Um acasalamento ruim de bases causa uma distorção da hélice de DNA duplo, o que impede que o nucleotídeo incorreto ocupe o local ativo da enzima.
3) Teste de leitura. A polimerase de DNA identifica nucleotídeos incorporados e elimina -os da cadeia filha. A atividade da exonuclease do DNA da polimerase quebra as ligações de fosfodiéster entre nucleotídeos na extremidade 3 'da nova cadeia.
Replicação de DNA em eucariotos
Ao contrário da replicação em procariontes, cuja replicação começa em um único site, a replicação eucariótica começa em vários locais de origem e o garfo de replicação move bidirecionalmente. Posteriormente, todos os garfos de replicação são mesclados, formando duas cromátides irmãs unidas no centrômero.
Eukaryotas tem muitos tipos de polimerase de DNA, cujos nomes usam letras gregas. Polimerase de DNA α formar um complexo com prima. Este complexo sintetiza iniciadores curtos que consistem em 10 nucleotídeos de RNA seguidos de 20 a 30 nucleotídeos de DNA.
Em seguida, DNA polimerase ε qualquer δ catalisa o alongamento da cadeia filha do primer. Polimerase de DNA ε Está envolvido na síntese da cadeia principal, enquanto a polimerase de DNA δ Sintetize a cadeia tardia.
Polimerase de DNA δ Prolongue o fragmento de Okazaki esquerdo até atingir o iniciador de RNA direito, produzindo um curto levantamento do primer ("abrete curto"). Ao contrário dos procariontes, onde um DNA da polimerase elimina o primer, nos eucariotos Uma enzima de retalho de endonuclease elimina o iniciador do RNA.
Em seguida, uma ligase de DNA sela os fragmentos de DNA que são adjacentes. A conclusão da replicação ocorre com a dissociação de proteínas do garfo de replicação.
O Replicação de DNA em eucariotos e ciclo celular
A replicação em eucariotos ocorre na fase S do ciclo celular. As moléculas de DNA replicadas são segregadas em duas células filhas durante a mitose. Fases G1 e G2 separam a fase S e mitose. A progressão através de cada fase do ciclo celular é altamente regulada por cinases, fosfatases e proteases.
Na fase G1 do ciclo celular, o complexo de reconhecimento de origem (OCR) se junta ao local de origem. Isso induz a união de helicas MCM e outras proteínas, como CDC6 e CDT1, para formar um complexo de pré-replicação (PRERC). Las Helicase McM se junta à cadeia de guias.
Pode atendê -lo: genes vinculadosNa fase S, o PRERC se torna um site de replicação ativo. As proteínas OCR, CDC6 e CDT1 são liberadas e a MCM Helicase se move na direção de 3 'a 5'. Quando a replicação terminar, isso será reiniciado no próximo ciclo celular.
Replicação das extremidades dos cromossomos em eucariotos
As extremidades dos cromossomos são conhecidas como telômeros, que consistem em sequências repetidas em conjunto, e de uma região de 3 'que se destaca, de 12 a 16 nucleotídeos de comprimento.
A polimerase de DNA é incapaz de replicar a extremidade de 3 'das cadeias de DNA. Isso ocorre porque a polimerase de DNA pode sintetizar apenas o DNA no endereço 5'-3 'e só pode prolongar as cadeias pré-existentes, sem poder sintetizar um primer nessa região. Consequentemente, os telômeros são reduzidos em cada rodada de replicação.
A enzima da telomerase impede o encurtamento dos telômeros. A telomerase é uma enzima que possui subunidades de proteínas e RNA (terceiro). Este último se liga às seqüências repetidas do DNA e permite que a telomerase se junte ao final do Telomer 3 '.
Uma sequência de RNA por trás da união localizada como um molde para a síntese de uma sequência de seis nucleotídeos (polimerização) no final da cadeia de DNA. O alongamento do telômer é catalisado por subunidades da telomerase, chamado transcriptase reversa de telomerase (TERT).
Após a polimerização, a translocalização ocorre, consistindo no movimento da telomerase para uma nova extremidade da cadeia de DNA, juntando seis outros nucleotídeos até o fim.
As funções de outras polimerases de DNA nos eucariotos
Polimerase de DNA β Tem um papel importante na eliminação de bases incorretas de DNA, mas não está envolvido na replicação do DNA.
Muitos descobriram o DNA da polimerase pertencente ao grupo de polimerase “replicação da translência”. Essas polimerases são responsáveis por sintetizar cadeias complementares em uma região de DNA danificada.
Existem vários tipos de polimerases "replicações de translesões". Por exemplo, DNA polimerase η Você pode replicar sobre Timina Dímeros, que são produzidos pela UV Light.
Replicação de DNA em Archeobacteria
A replicação do DNA do Archeobacteria é semelhante à dada em eucariotos. Isso se deve ao seguinte: 1) proteínas que participam da replicação são mais parecidas com as eucariotas do que as dos procariontes; e 2) embora exista apenas um local de replicação como nos procariontes, sua sequência é semelhante ao local de origem eucariótica.
A semelhança na replicação entre os arcos e os eucariotos apóia a idéia de que ambos os grupos são filogeneticamente mais relacionados entre si do que qualquer um deles com os procariontes.
Referências
- Brooker, r. J. 2018. Análise e princípios genéticos. McGraw-Hill, Nova York.
- Hartwell, l. H., Goldberg, m. eu., Fischer, J. PARA., Hood, l. 2018. Genética - de genes genomas. McGraw-Hill, Nova York.
- Kušić-Tišma, j. 2011. Aspectos fundamentais da replicação do DNA. Acesso aberto Intech, Croácia.
- Lewis, r., 2015. Conceitos e aplicações de genética humana. McGraw-Hill, Nova York.
- Pierce, b. PARA. 2005. Genética - uma abordagem conceitual. C. H. Freeman, Nova York.

