Tradução do processo de DNA em eucariotas e procariotos
- 4371
- 1229
- Pete Wuckert
O Tradução de DNA É o processo pelo qual as informações contidas nos mensageiros produzidos durante a transcrição.
Do ponto de vista celular, a expressão de um gene é uma questão relativamente complexa que ocorre em duas etapas: transcrição e tradução.
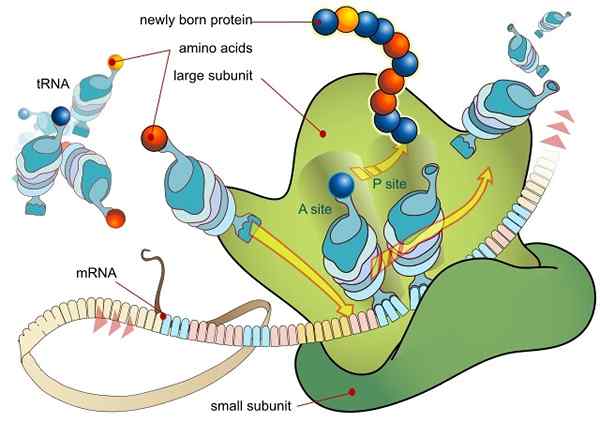
 Tradução de RNA mediada por um ribossomo (Fonte: Ladyofhats / Domínio Público, via Wikimedia Commons)
Tradução de RNA mediada por um ribossomo (Fonte: Ladyofhats / Domínio Público, via Wikimedia Commons) Todos os genes que são expressos (codificando ou não sequências peptídicas, ou seja, proteínas) o fazem inicialmente transferindo as informações contidas em sua sequência de DNA para uma molécula de Mer Mensageiro (RNAM) através de um processo chamado transcrição.
A transcrição é obtida por enzimas especiais conhecidas como RNA polimerases, que usam uma das cadeias complementares do DNA do gene como um molde para a síntese de uma molécula de "pré-arnm", que é posteriormente processada para formar uma madatura madura.
Para genes que codificam proteínas, as informações contidas no RNM maduro são "lidas" e traduzidas em aminoácidos de acordo com o código genético, que especifica qual códon ou triplo de nucleotídeo corresponde a cada aminoácido em particular.
A especificação da sequência de aminoácidos de uma proteína, portanto, depende da sequência inicial de bases de nitrogênio no DNA que corresponde ao gene e, em seguida, no RNA que transporta as informações do núcleo para o citosol (nas células eucarióticas); processo que também é definido como a síntese de proteína guiada por RNM.
Em vista do fato de que existem 64 combinações possíveis das 4 bases de nitrogênio que formam DNA e RNA e apenas 20 aminoácidos, um aminoácido pode ser codificado por diferentes trigêmeos (códons), portanto, diz -se que o código genético é " Degenerado "(exceto a meão de aminoácidos, que é codificada por um códon AUG exclusivo).
[TOC]
Tradução Eukaryota (etapas-processos)
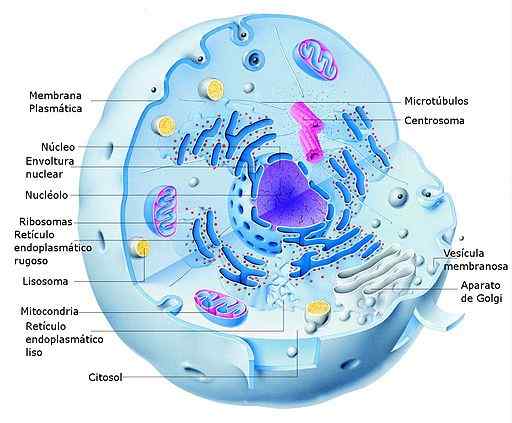
 Diagrama de uma célula de eukaryot animal e suas partes (fonte: Alejandro Porto [CC0] via Wikimedia Commons)
Diagrama de uma célula de eukaryot animal e suas partes (fonte: Alejandro Porto [CC0] via Wikimedia Commons) Nas células eucarióticas, a transcrição ocorre no núcleo e na tradução para o citosol, de modo que os RNMs que são formados durante o primeiro processo também cumprem uma função no transporte das informações do núcleo para o citosol, onde o maquinário biossintético (ribossomos ).
É importante mencionar que a compartimentalização da transcrição e tradução nos eucariotos é verdadeira para o núcleo, mas não é o mesmo para as organelas com seu próprio genoma, como cloroplastos e mitocôndrias, que têm sistemas mais semelhantes aos de organismos procarsoóticos.
As células eucarióticas também apresentam ribossomos citosólicos ligados às membranas do retículo endoplasmático (retículo endoplasmático endoplasmático áspero), no qual a tradução das proteínas que se destina a serem inseridas nas membranas celulares ou que requerem o processamento pós -translacional que ocorrem no referido condimento oculto.
- Processamento RNM antes de sua tradução
Os RNMs são modificados em suas extremidades à medida que são transcritas:
- Quando a extremidade 5 'do RNM emerge da superfície da RNA polimerase II durante a transcrição, isso é "atacado" imediatamente por um grupo de enzimas que sintetizam um "capô" composto por 7-metilunilado e que está conectado ao terminal nucleotídeo do RNM através de um link de 5 'tiffosfato, 5'.
Pode atendê -lo: códon- O final de 3 'do RNM sofre um "clivaje" por uma endonuclease, que gera um grupo hidroxila livre livre que se une a um "ristra" ou "cauda" de resíduos de adenina (de 100 a 250) que são adicionados no mesmo Hora de uma enzima Poli (A) Polimerase.
O "capô 5 '" e o "cauda Poli A ”Eles cumprem as funções na proteção de moléculas de RNM contra a degradação e, além disso, trabalham no transporte de transcrições maduras para o citosol e no início e rescisão da tradução, respectivamente.
COrte e Empalme
Após a transcrição, os RNMs “primários” com seus dois extremos modificados, ainda presentes no núcleo, passam por um processo de “corte e splicing” pelo qual as sequências intronas são geralmente eliminadas e os exons resultantes são unidos (processamento pós -regional), com o que as transcrições maduras que abandonam o núcleo e alcançam o citosol são obtidas.
O corte e a emenda são realizados por um complexo riboproteico chamado Esplicleosoma (Anglicismo de Spliceossoma), formado por cinco pequenas ribonucleoproteínas e moléculas de RNA, que são capazes de "reconhecer" as regiões que devem ser eliminadas da transcrição primária.
Em muitos eucariotos, existe um fenômeno conhecido como "corte e articulação alternativa", o que significa que diferentes tipos de modificações pós -registracionais podem causar proteínas diferentes ou isoenzimas que diferem entre si em alguns aspectos de suas seqüências.
- Os ribossomos
Quando as transcrições maduras deixam o núcleo e são transportadas para tradução para o citosol, estes são processados pelo complexo de tradução conhecido como ribossomo, que consiste em um complexo proteico associado a moléculas de RNA.

Os ribossomos são compostos de duas subunidades, uma "grande" e outra "pequena", que são dissociadas livremente no citosol e se juntam ou se associam na molécula de mRNA que traduz.
A união entre os ribossomos e o mRNA depende de moléculas de RNA especializadas que estão associadas a proteínas ribossômicas (RNA ribossômico ou RNA e transferência ou RNA ARNT), cada um dos quais exerce funções específicas.
Os ARNT são "adaptadores" moleculares, porque, através de um de seus fins, eles podem "ler" cada códon ou trigêmeo no RNA maduro (por complementaridade das bases) e, através do outro, eles podem se juntar ao aminoácido codificado pelo códon "Read".
As moléculas de RNR, por outro lado, são responsáveis por acelerar (catalisar) o processo de ligação de cada aminoácido na cadeia peptídica nascente.
Um RNM maduro eucariótico pode ser "lido" por muitos ribossomos, tantas vezes quanto a célula indica. Em outras palavras, o mesmo RNM pode levar a muitas cópias da mesma proteína.
Iniciando o códon e o quadro de leitura
Quando um RNM maduro é abordado pelas subunidades ribossômicas, o complexo riboproteico "escane" a sequência da referida molécula até que um códon inicial encontre, que é sempre agosto e implica a introdução de um resíduo de metionina.
Pode servir a você: monoplóide: como isso acontece, organismos, frequência e utilidadeO códon AUG define o quadro de leitura para cada gene e, além disso, define o primeiro aminoácido de todas as proteínas traduzidas para a natureza (esse aminoácido é frequentemente eliminado pós -translacionalmente).
Códons de rescisão
Três outros códons foram identificados como aqueles que induzem o término da tradução: UAA, UAG e UGA.
Essas mutações que implicam uma mudança de bases de nitrogênio no trigêmeo que codifica para um aminoácido e que resultam em códons de rescisão são conhecidos como mutações sem sentido, pois causam detenção prematura do processo de síntese, que forma proteínas mais curtas mais curtas.
Regiões não traduzidas
Perto da extremidade 5 'das moléculas RNM maduras, existem regiões que não são traduzidas (UTR, do inglês Região não traduzida), também chamado de sequências "líderes", localizadas entre o primeiro nucleotídeo e o início da tradução (agosto).
Essas regiões da UTR que não são traduzidas têm locais específicos para união com ribossomos e humanos, por exemplo, eles têm um comprimento aproximado de 170 nucleotídeos, entre os quais existem regiões regulatórias, locais de ligação a proteínas que funcionam na regulação da tradução etc.
- Início da tradução
A tradução, bem como a transcrição, consiste em 3 fases: uma de iniciação, outra de alongamento e, finalmente, uma de terminação.
Iniciação
Consiste na montagem do complexo translacional no RNM, que merece a união de três proteínas conhecidas como fatores de iniciação (se, de inglês Fator de iniciação) Se1, if2 e if3 para a pequena subunidade do ribossomo.
O complexo "pré -initação" formado pelos fatores de iniciação e a pequena subunidade ribossômica são unidos, por sua vez, com um ARNT que "carrega" um resíduo metionina e esse conjunto de moléculas se liga ao RNAM, perto do códon inicial Aug.
Esses eventos levam à união RNM com a grande subunidade ribossômica, o que leva à liberação de fatores de iniciação. A grande subunidade do ribossomo possui 3 locais de união para moléculas de ARNT: Local A (aminoácido), Local P (Polipeptídeo) e Local E (saída).
O local A se junta ao anticod aminoacil-Antinte, que é complementar ao do mRNA que é traduzido; O local P é onde o aminoácido é transferido do peptídeo arnt para o local nascente e o local S é onde está em arnt "vazio" antes de ser liberado para o citosol depois de entregar o aminoácido.
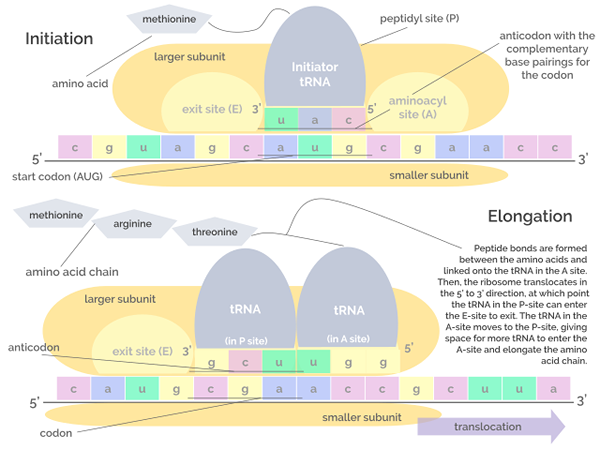 Representação gráfica das fases de iniciação e alongamento da tradução (Fonte: Jordan Nguyen/CC BY-S (https: // CreativeCommons.Org/licenças/BY-SA/4.0) via Wikimedia Commons)
Representação gráfica das fases de iniciação e alongamento da tradução (Fonte: Jordan Nguyen/CC BY-S (https: // CreativeCommons.Org/licenças/BY-SA/4.0) via Wikimedia Commons) Alongamento
Esta fase consiste no "movimento" do ribossomo ao longo da molécula de mRNA e na tradução de cada códon que "lendo" o que implica o crescimento ou alongamento da cadeia polipeptídica no nascimento.
Esse processo requer um fator conhecido como fator de alongamento do GTP na forma de GTP, que é o que impulsiona a translocação de fatores de alongamento ao longo da molécula RNM enquanto está traduzindo.
Pode atendê -lo: fragmentos okazakiA atividade peptidil da transferase dos RNAs ribossômicos permite a formação de ligações peptídicas entre aminoácidos sucessivos que são adicionados à cadeia.
Terminação
A tradução termina quando o ribossomo atende a qualquer um dos códons de rescisão, uma vez que os ARNs não reconhecem esses códons (eles não codificam aminoácidos). Proteínas conhecidas como fatores de liberação também são unidas, o que facilita o destacamento do Ribosoma MR.
Tradução procaria (passagens-processos)
Nos procariontes, como nas células eucarióticas, os ribossomos encarregados da síntese de proteínas são encontrados no citosol (que também é verdadeiro para máquinas transcricionais), fato que permite o rápido aumento na concentração citosólica de uma proteína quando a expressão dos genes que A codifica aumenta.
Embora não seja um processo extremamente comum nesses organismos, o RNM primário produzido durante a transcrição pode sofrer a maturação pós -registracional por meio de "corte e splicing". No entanto, o mais comum é observar os ribossomos ligados ao primário transcrito que o traduzem ao mesmo tempo em que está sendo transcrito da sequência de DNA correspondente.
Em vista do exposto, a tradução em muitos procariontes começa com o final 5 ', uma vez que o final do mRNA de 3' permanece ligado ao DNA do molde (e ocorre concomitantemente com a transcrição).
Regiões não traduzidas
As células procarióticas também produzem RNM com regiões não translatadas que são conhecidas como "caixa de brilho-Dalgarno" e cuja sequência de consenso é Aggaggg. Como é evidente, as regiões da UTR de bactérias são consideravelmente mais curtas que as das células eucarióticas, embora exerçam funções semelhantes durante a tradução.
Processo
Nas bactérias e outros organismos procarióticos, o processo de tradução é bastante semelhante ao das células eucarióticas. Também consiste em três fases: iniciação, alongamento e rescisão, que dependem de fatores procarióticos específicos, diferentes dos usados pelos eucariotos.
O alongamento, por exemplo, depende de fatores de alongamento conhecidos como EF-Tu e EF-Ts, em vez do fator eucariótico G.
Referências
- Alberts, b., Johnson, a., Lewis, J., Raff, m., Roberts, k., & Walter, P. (2007). Biologia da célula molecular. Garland Science. Nova York, 1392.
- Clay, s. & Brown, W. (2008) Tradução: DNA para mRNA para proteína. Nature Education 1 (1): 101.
- Griffiths, a. J., Wessler, s. R., Lewontin, r. C., Gelbart, w. M., Suzuki, d. T., & Miller, J. H. (2005). Uma introdução à análise genética. Macmillan.
- Lodish, h., Berk, a., Kaiser, c. PARA., Krieger, m., Scott, m. P., Bretscher, a.,… & Matsudaira, P. (2008). Biologia celular molecular. Macmillan.
- Nelson, d. eu., Lehninger, a. eu., & Cox, M. M. (2008). Lehninger Principles of Biochemistry. Macmillan.
- Rosenberg, l. E., & Rosenberg, D. D. (2012). Genes e genomas humanos: ciência. Saúde, Sociedade, 317-338.
- « Características de Sorbus ária, habitat, propriedades, cultura
- Boletus aereus características, habitat, identificação, receitas »

